Serviços Personalizados
Artigo
Indicadores
Links relacionados
 Citado por SciELO
Citado por SciELO
 Similares em SciELO
Similares em SciELO
Bookmark
Visión Científica
versão impressa ISSN 2222-4361
Vis cienti. v.1 n.2 La Paz 2009
ARTICULO
DETECCIÓN DEL MARCADOR Y-STR DE USO FORENSE DYS393 EN HECES HUMANAS
FUENTES B. Susy, CONDORI T. Gualberto, LUNA B Ruddy,
Centro de Investigación Genética, DNPTC-Policía Nacional, Bolivia. Correspondencia: rlunab@gmail.com
Mediante la amplificación por PCR del marcador molecular de uso forense DYS393, se ha evaluado cualitativamente el sistema comercial “Fecal DNA Purification Kit” de la compañía MoBio, para la purificación de ADN total a partir de muestras de heces de origen humano, conservadas a -20 ºC por 6, 13 y 90 días, sometidas a degradación ambiental por 24 horas, y colectadas en vía pública. Los resultados demostraron que el sistema es eficiente en todos los casos, tanto para remover los inhibidores de PCR como para obtener cantidad suficiente del disminuido material genético humano remanente en los restos fecales, y así permitir la detección y estudio forense del marcador de identidad genética. Un siguiente estudio cuantitativo es requerido.
Using the genetic identity marker DYS393 of forensic use, by means of PCR it has been evaluated the commercial system “Fecal DNA Purification Kit” from MoBio company for the purification of total DNA, starting from samples of recent grounds, subjected to environmental degradation along 24 hours, preserved at -20ºC, and sampled from thoroughfare. Results showed that the system is efficient in all cases, so much to remove the inhibitors of PCR like to obtain enough quantity of the diminished and remnant human genetic material in the fecal samples, reaching the goal of detecting this genetic identity molecular marker in prospective forensic applications. A quantitative analysis is required.
Palabras clave: Heces, evidencia, forense, purificación, perfil genético, DYS393.
INTRODUCCIÓN
En las pasadas tres décadas, el progreso científico en el desarrollo de técnicas de análisis genético molecular ha sido vertiginoso. Para las ciencias forenses, la incorporación del perfil de identidad genética como herramienta en la identificación de personas representa uno de los avances más útiles y significativos desde la creación de las bases de datos con huellas dactilares.
Diversos marcadores moleculares del tipo STR (Single Tandem Repeats) son empleados actualmente para obtener perfiles de identidad genética; uno de ellos, el marcador DYS393, que se halla únicamente en el cromosoma Y, es principalmente utilizado para discriminar, en uso conjunto a otros marcadores similares, el perfil genético del ADN autosómico de una víctima de sexo femenino, del perfil haplotípico de un agresor varón, en un caso típico de agresión sexual que contiene evidencia de indicios biológicos mezclados.
Sin embargo, en un escenario criminal puede encontrarse una amplia gama de indicios biológicos durante una investigación policial forense, entre los que se incluyen restos titulares, manchas hemáticas, muestras pilosas, fluidos diversos, y, rara vez, restos fecales. Este tipo último de indicio, por su naturaleza, es difícil de procesar en el laboratorio, especialmente si la descomposición orgánica ha sido acelerada por condiciones de temperatura y humedad del entorno, y si el tiempo transcurrido ha sido excesivo. ¿Cuál entonces la razón de evaluar métodos analíticos para estudiar esta rara evidencia biológica? Pues como ejemplo y motivo principal está el atraco a la casa de cambios SUDAMER en La Paz en 2006, donde se obtuvo como único rastro biológico este singular pero escaso tipo de evidencia (com.pers. Tcnl. Gualberto Condori T., Sub-Jefe Dpto. Nal. Policía Técnica Científica).
Existen tres razones principales por las que la obtención de ADN útil a partir de heces es compleja: a) la cantidad de material genético humano es escasa; b) existe una gran cantidad de material genético microbiano contaminante; y c) las heces poseen una variedad de compuestos inhibidores del PCR como las bilirrubinas, polisacáridos complejos, sales biliares y otros (Deuter et al., 1995; Monteiro et al., 1997).
La facultad para obtener perfiles de identidad genética a partir de muestras de heces, resulta ventajosa bajo circunstancias en las que una investigación policial forense dispone únicamente de este material biológico como evidencia; un método rápido y versátil de purificación resulta, por tanto, muy necesario (Vigilant, 2002). La actividad inhibitoria de los contaminantes propios del material fecal ha sido previamente documentada (Surace, 1999; Monteiro et al., 1977). Esta inhibición obstaculiza principalmente la actividad de la enzima polimerasa durante la amplificación por PCR de los marcadores STR, por lo que su remoción o inactivación es imperativa. (Deuter et al., 1995). Los factores metodológicos más importantes ha tomar en cuenta son: la etapa de purificación durante la obtención del material genético total, la inactivación de inhibidores durante la lisis celular y la purificación durante la recuperación de la cromatina nuclear de alto peso molecular que contiene el genoma del cromosoma Y.
La aplicación de varios protocolos de purificación de ADN de rutina han mostrado en nuestro medio poca o ninguna eficiencia con este tipo específico de evidencia biológica (com.pers. Tcnl. Condori), por lo que se hace necesario evaluar nuevos y mejor elaborados sistemas, basados en mecanismos de purificación más selectiva y eficiente tal como ha sido demostrado por el trabajo de Vandenberg y van Oorschot (2002) con un método de purificación de la compañía QIAGEN.
La sola detección del marcador DYS393 en heces fecales humanas mediante PCR es un indicador cualitativo de la eficiencia del método de purificación propuesto, así como también de la sensibilidad del sistema de amplificación (considerando la disminuida cantidad de ADN del donante y el efecto degradativo de sustancias y microorganismos propios de las heces), y, adicionalmente, es un indicador directo del sexo del donante de la muestra biológica fecal.
El presente trabajo evaluó el sistema comercial “Fecal DNA Purification Kit” de lisis alcalina y filtrado selectivo por columnas de precipitación de la compañía MoBio (diseñado originalmente para estudiar el genoma total bacteriano), en búsqueda de la posibilidad de extender su aplicabilidad en el campo forense, determinando si posee la sensibilidad suficiente para detectar el casi imperceptible ADN humano presente en restos fecales, a través de la amplificación y detección del microsatélite DYS393.
MATERIALES Y MÉTODOS
Dos tipos de muestras fecales fueron obtenidas para el estudio. La primera constituida por heces fecales humanas de donante varón, colectadas en frascos plásticos inmediatamente a su deposición. La segunda de heces humanas de donante desconocido colectadas en vía pública.
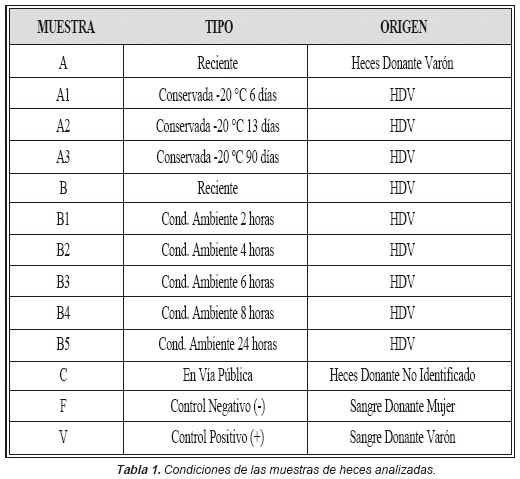
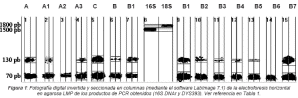
Para estimar cualitativamente la eficiencia del método aplicado, se previó la purificación de ADN a partir de la una muestra principal colectada y procesada fresca etiquetada “A”. De ésta, tres sub-muestras fueron conservadas a -20 ºC y procesadas al término de 6 días (“A1”), de 13 días (“A2”) y de 90 días (“A3”) de conservación respectivamente. Una cuarta sub-muestra de A, etiquetada “B” (por tanto réplica de A), fue sometida a condición ambiente por 24 horas, obteniendo fragmentos de la misma cada 2 horas (B1, B2, B3, B4 Y B5). La muestra obtenida en vía pública fue etiquetada “C”.
El control negativo de donante femenino fue etiquetado “F” y el control positivo “V”.
Los extractos fueron primeramente sometidos a un PCR buscando detectar un marcador propio y exclusivo de microorganismos bacterianos (el gen de la sub-unidad 16S del ribosoma, 1500 pb aprox.) con objeto de evaluar la pureza e idoneidad del extracto para futuras amplificación a partir del ADN total obtenido. Posteriormente se procedió a la detección del marcador DYS393 del genoma humano (130 pb aprox.).
La especificidad de los cebadores se confirmó utilizando ADN control negativo de un donante femenino (“F”), y ADN de un donante varón como control positivo (“V”), obtenidos a partir de sangre colectada por punción y goteo (50 ul aprox.).
Se mantuvo el esquema general recomendado por el fabricante aplicando algunas modificaciones: 400 a 500 mg de heces fueron agregados a un tubo MoBio de miniesferas de sílica, adicionando 700 ul de la solución para mini esferas más 250 ul de la solución removedora de contaminantes (Inhibitor Removal Solution, IRS). La mezcla se sometió a vórtex por 10 minutos. Luego los tubos fueron centrifugados a 10000G por 1 minuto y se colectó el sobrenadante en un tubo eppendorf nuevo, adicionando 250 ul de la solución de lisis, dejando en reposo a 4 ºC por 10 minutos luego de mezclarlos por inversión. Posteriormente se colectaron 600 ul de la mezcla en un tubo filtro spin, centrifugando el mismo por 1 minuto a 10000G y desechandose el precipitado. El remanente de la mezcla fue sometido al mismo procedimiento, desechando el precipitado y se agregando al filtro spin 600 ul de una solución de etanol al 90%. El filtro spin con etanol fue centrifugado a 10000G por 1 minuto, desechado el precipitado y se centrifugando una vez más por 1 minuto a 10000G. Finalmente, el filtro spin fue colocado en un tubo eppendorf nuevo y se agregaron 50 ul de una solución Tris 10 mM, precipitando y recuperando el ADN total (> 50 kDa) por centrifugación de 1 minuto a 10000 G.
Se utilizó el mismo sistema pero eliminado la etapa inicial de disgregación por miniesferas, más la de remoción de contaminantes con la solución IRS: 50 ul de sangre fresca fueron colocados en un tubo eppendorf de 1.5 ml y se agregaron 800 ul de la solución de lisis alcalina, dejando reposar durante 20 minutos a 4 º C luego de mezclar por inversión. Se colectaron 600 ul de la mezcla en un tubo filtro spin que fue centrifugado por 1 minuto a 10000G, desechando el precipitado. El remanente de la mezcla fue sometido al mismo procedimiento. Desechando el filtrado, se agregaron 600 ul de la solución de etanol al 90% y se centrifugó a 10000G por 1 minuto, el precipitado fue desechado y se centrifugó una vez más por 1 minuto a 10000G. El filtro spin fue colocado en un tubo nuevo y se agregó cuidadosamente 50 ul de Tris 10 mM, liberando el ADN del filtro finalmente por centrifugación de 1 minuto a 10000 G.
La amplificación del 16S DNA ribosomal para detectar genoma bacteriano presente en las heces se realizó según el protocolo estandarizado para el estudio de bacterias sulfato reductoras en sedimentos amazónicos por Luna R. (2004).
Las condiciones de los reactivos BioLabs, cuyo Buffer contiene MgCl2 para la amplificación del marcador humano DYS393, fueron: Buffer Taq 1X, dNTP´s 0.2mM, Ceba-dores 0.2uM, Taq Polimerasa 0.035 U/ul y de 3 a 5 ng del ADN purificado. El programa de amplificación empleado fue: un ciclo de 95 ºC por 1 minuto; 30 ciclos de 95 ºC por 30 segundos, 55 ºC por 30 segundos y 72 ºC por 30 segundos; 1 ciclo final de 72 ºC por 5 minutos (Lisbeth Borjas, Universidad del Zulia, Venezuela. com. pers.).
Los purificados del ADN total obtenido y el amplificado del 16S DNAr bacteriano fueron revelados mediante electroforesis horizontal en agarosa estándar al 2%, 50V y 45 minutos. El producto DYS393 fue revelado por electroforesis horizontal en agarosa LMP al 2.5%, 50V y 30 minutos. Las fotografías digitales fueron analizadas empleando el programa informático de distribución libre LabImage 7.1.
RESULTADOS
La purificación de ADN en los grupos de muestras A y B permitió obtener de 15 a 20 ug/ml de material genético. La muestra C proporcionó 3 a 7 ug/ml y presentó una degradación aparente entre 82 y 93%, evaluada por comparación del esmirro y la longitud total de la corrida electroforética mediante el programa LabImage. En todos los casos se obtuvo el producto esperado de 1500 pb correspondiente al 16S DNAr bacteriano (no mostrado), y en todos, excepto en el control negativo F, se obtuvo el amplificado esperado de 130 pb del DYS393 (Figura 1).
DISCUSIONES Y CONCLUSIONES
La obtención de perfiles de ADN a partir de heces se ha vuelto mucho más efectiva y práctica con los sistemas de extracción automatizados (Norris y Bock, 2000; Roy, 2003), pero son mucho más costosos que los manuales, y mucho más (hasta 20 veces) que los protocolos tradicionales. La compañía MoBio ofrece un “kit” manual específico para aplicaciones forenses pero con un costo 5 veces mayor al del “kit” evaluado en el presente trabajo. Convenientemente, los resultados experimentales obtenidos en el presente trabajo han demostrado eficiencia suficiente para proseguir con una estandarización.
Un sistema de lisis únicamente alcalino es levemente selectivo ya que al no contener SDS limita la lisis celular en su mayor parte a membranas, dejando paredes celulares pobremente afectadas. Esto es apropiado en muestras de heces ya que la mayor parte de los ácidos nucleícos presentes pertenecen a bacterias de la flora intestinal y otros contaminantes biológicos parasíticos.
El método empleado de MoBio contiene SDS como detergente lísico y, pese a esto, la potencial interferencia del genoma bacteriano en el PCR ha sido evadida por la especificidad del marcador STR elegido. No se obtuvieron productos adicionales al esperado a excepción de la banda extrema inferior de aproximadamente 70 pb, presente en todos amplificados y asociada a la naturaleza del DYS393 y su probable afinidad a regiones del cromosoma X (Neil et al., 2004) que no afectan sueficiencia para detectar el producto esperado en el cromosoma Y de 130pb.
La cantidad de ADN humano presente en las muestras analizadas no puede ser apropiadamente evaluada con los métodos empleados, pero, a través de los resultados obtenidos, ha demostrado ser de calidad y cantidad suficientes para realizar la amplificación del marcador forense DYS393 a partir de muestras fecales en distintos estados de conservación (A, B y C). Mientras mayor el tiempo de reposo de restos fecales a condiciones ambientales, mayor el estado degradativo de sus componentes biológicos. Los restos del epitelio intestinal arrastrados durante la formación y expulsión de las heces son mínimos y generalmente degradados por la actividad microbiana, pese a esto la cantidad detectada por el sistema empleado ha sido suficiente, lo cual invita a continuar la investigación utilizando métodos cuantitativos.
La presencia del producto 16S DNAr es evidencia de la óptima calidad del material genético para el PCR (1500 pb). La gran cantidad de bacterias presentes en heces probablemente facilita su detección, pero la presencia de contaminantes orgánicos e inhibidores dificultarían la misma al emplearse métodos convencionales de extracción de ADN (Wilson, 1997). El protocolo empleado no solo ha permitido la amplificación de un marcador taxonómico bacteriano, sino también de uno humano y de extenso uso forense.
La ausencia de producto DYS393 en el carril 3 (muestra congelada por 13 días) no implica un efecto negativo del tiempo de conservación sobre la integridad del ADN, ya que en una prueba posterior la réplica de esta muestra evidenció un resultado positivo (datos no mostrados).
Sin embargo, el ensayo realizado con la muestra B (condición ambiente durante 24 horas) deja ver una gradual disminución de intensidad de las bandas en la electroforesis, lo cual puede asociarse al probable efecto degradativo del entorno sobre el ADN humano remanente en las heces. No obstante, al tratarse de un PCR no cuantitativo no es posible llegar a una clara conclusión. Para apoyar este punto de vista nótese la similitud de intensidad y tamaño entre las bandas del control positivo V (carril 15) y de la muestra colectada en vía pública (carril 5).
Un resultado adicional deducido a partir de la presencia de DYS393 en la muestra C (carril 5) es la posibilidad de afirmar que se trató de heces procedentes de un donante varón. Lo cual evidencia la potencial aplicación de los marcadores del cromosoma-Y en este tipo de evidencia biológica para, inicialmente, identificar el género del donante, además de evidenciar claramente la importante posibilidad de estudiar el genoma humano presente en una muestra tan maltratada.
El principio de remoción de inhibidores de PCR ha sido previamente demostrado en aplicaciones forenses (Norris y Bock, 2000; Johnson et al., 2005).
La eficiencia demostrada por el método aplicado tiene directa relación con los principios físico-químicos del sistema comercial utilizado (MoBio). Por un lado, una optimización metodológica en la aplicación de la solución removedora de contaminantes “IRS”, y por otro, la purificación final del ADN obtenido a través del filtro “spin” son, al punto de vista de los autores, los factores preponderantes para lograr resultados positivos con este tipo de muestra biológica compleja, considerada como potencial evidencia biológica durante una investigación forense, que por su naturaleza se ve sometida a descomposición por condiciones ambientales extremas y por la actividad bacteriana destructiva inherente.
AGRADECIMIENTOS
A Susana Revollo PhD, responsable del laboratorio de Biología Molecular del SELADIS en La Paz por el soporte técnico prestado.
REFERENCIAS BIBLIOGRÁFICAS
1. Deuter R, Pietsch S, Hertel S, Muller O. A method for preparation of fecal DNA suitable for PCR. Nucleic Acids Research 1995. 23(18):3800-1. [ Links ]
2. Johnson D, Martil L. y Roberts K. STR-typing of human DNA from human fecal matter using the QIAGEN QIAamp® stool mini kit. Journal of Forensic Sciences 2005. 50:4.
3. Monteiro L, Bonnemaison D, Vekris A, Petry KG, Bonnet J. 1997. Complex polysaccharides as PCR inhibitors feces. Journal of Clinical Microbiology. 1997:995-998.
4. Nicholas Vandenberg y Roland A. H. van Oorschot. Extraction of Human Nuclear DNA from Feces Samples Using the QIAamp DNA Stool Mini Kit. Journal of Forensic Sciences 2002, 47(5):1-3.
5. Leat Neil, Liezle Ehrenreich, Mongi Benjeddou y Sean Davison. Developments in the use of Y- chromosome markers in forensic genetics. African Journal of Biotechnology 2004 . 3 (12): 637-642.
6. Luna R. Perfil preliminar de bacterias sulfato reductoras en sedimentos de laguna “La Granja” (Beni - Bolivia). Tesis de Licenciatura. Carrera de Biología, FCPN, UMSA, La Paz, Bolivia 2004.Norris D. y Bock J. Use of fecal material to associate a suspect with a crime scene: report of two cases.Journal of Forensic Sciencies 2000. 45:1.
7. Surace D. Retrieval of nuclear DNA from human feces:Implications and techniques in forensic science [hons. thesis]. La Trobe University 1999. [ Links ]
8. Taberlet Pierre, Sally Griffin, Benoît Goossens, Sophie Questiau, Valérie Manceau, Nathalie Escaravage, Lisette P. Waits and Jean Bouvet. Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic Acids Research1996. 24(16):3189-3194.
9. Linda Vigilant. Technical Challenges in the Microsatellite Genotyping of a Wild Chimpanzee Population Using Feces. Evolutionary Anthropology, Suppl 2002. 1:162- 16.
10. Ian G. Wilson, Inhibition and Facilitation of Nucleic Acid Amplification. Applied and Environmental Microbiology 1997. 63(10):3741-3751. [ Links ]
11. De La Barra Zeballos Susan, Identificación de anticuerpos anticélulas de los islotes del páncreas (ICAs) en pacientes con diabetes Mellitus tipo 1 y en familiares de primer grado de los mismos por inmunofluorescencia indirecta con sustrato de páncreas de rata, Tesina para optar al título de Licenciatura. UMSA - FCFB. La Paz Bolivia. 2000. [ Links ]
12. Díaz Horta O, Cabrera Rode E, Díaz Díaz O. Estrategias a seguir en la prevención primaria de Diabetes Mellitus Insulinodependiente. Revista Cubana Endocrinología, 1996;7(1) 12-25. [ Links ]
13. Sánchez-García L, Salinas MT. Un modelo de aplicación de la medicina preventiva: Diabetes Mellitus Insulino Dependiente. SELADIS - FCFB y Hospital Obrero CNS - Medicina Interna y Endocrinología. La Paz - Bolivia. 1997. [ Links ]