Serviços Personalizados
Artigo
Indicadores
Links relacionados
 Citado por SciELO
Citado por SciELO
 Similares em SciELO
Similares em SciELO
Bookmark
Visión Científica
versão impressa ISSN 2222-4361
Vis cienti. v.1 n.2 La Paz 2009
ARTICULO
IDENTIFICACIÓN DE SALMONELLA SP MEDIANTE LA TÉCNICA DE REACCIÓN EN CADENA DE LA POLIMERASA ANIDADO (NESTED PCR) Y TÉCNICAS CONVENCIONALES EN HUEVOS RECOLECTADOS EN LOS PRINCIPALES MERCADOS DE LA CIUDAD DE LA PAZ
ESPINOZA Edy1; REVOLLO Susana1; ESPADA Angélica2;
1 Laboratorio de Biología Molecular, Instituto SELADIS;
2 Laboratorio de Microbiología de Alimentos, Instituto SELADIS
* Correspondencia autor: edy_espinoz@hotmail.com.
RESUMEN
La salmonelosis es una enfermedad de distribución mundial, siendo frecuntes los brotes de enfermedades transmitidas por alimentos contaminados. El 2001 en Bolivia, del total de casos reportados a nivel nacinal de infección por Salmonella, el 47% fueron positivos, de acuerdo al informe del Sistema Nacional de Información en Salud (SNIS), Servicio Departamental de Salud, Ministerio de Salud y Previsión Social. En el presente estudio se analizó 40 muestras de huevos procedente de diferentes mercados de la ciudad de La Paz mediante cultivos bacteriológicos y la técnica de reacción en cadena de la polimerasa anidado (Nested-PCR). Se identificó la sencibilidad y especificidad del método, comparada con la técnica convencional, para su posterior aplicación.
ABSTRACT
Salmonelosis is considered a disease of world distribution. Contaminated foods constitute the main source of infection. In 2001 in Bolivia, 47% of the reported cases were positive, according to the Sistema Nacional de Información en Salud (SNIS), Servicio Departamental de Salud, Ministerio de Salud y Previsión Social. In the present work samples consisted on forty eggs were analyzed, collected from different markets of La Paz city. The identification of Salmonella was established by bacteriological culture and Nested Polymerase chain reaction (Nested PCR). Salmonella sp was identified in 17.5 % of the studied samples by Nested PCR. This technique shows higher sensitivity, specificity and efficiency compared to the traditional bacteriological culture method.
Palabras Clave:Nested PCR, Primer
INTRODUCCIÓN
La salmonelosis es una enfermedad de distribución mundial, siendo frecuentes los brotes de enfermedades transmitidas por alimentos contaminados o por diseminación proveniente de un portador desconocido. La infección por salmonella es un problema importante de salud pública, tanto en países desarrollados como subdesarrollados. (3)
Según el informe mensual del SNIS (Sistema Nacional de Información en Salud) del SEDES (Servicio Departamental de Salud), en el ámbito urbano/rural de Bolivia, del total de casos reportados a nivel nacional de infección por Salmonella, el 47 % fueron positivos en el año 2001. (23)
Todas las Salmonellas deberían ser consideradas como potencialmente patógenas para el hombre. La única vía de entrada de estos microorganismos en el cuerpo humano es la oral, por lo que es de suma importancia el análisis de los alimentos. (13)
Los miembros del género Salmonella son los agentes causales de diferentes infecciones intestinales, entre éstas la salmonelosis humana, y pueden dividirse en los siguientes grupos: Las fiebres entéricas (fiebre tifoidea), bacteriemias en lesiones focales y enterocolitis o gastroenteritis. (6)
En una infección por alimentos debida a Salmonella, los síntomas sólo aparecen cuando el patógeno se multiplica en el intestino. Su período de incubación es variable según la especie, una vez ingerido el alimento. (5)
El avance en la biotecnología ha permitido el desarrollo de técnicas moleculares para el estudio del genoma de Salmonella. Los primeros resultados manifiestan la presencia de secuencias de inserción. Asimismo, estos estudios han permitido diseñar primers específicos de estas regiones y poder, utilizarlos en la identificación especifica de Salmonella a través de métodos de amplificación como ser la Reacción en Cadena de Polimeraja en (PCR). (8)
En el presente estudio se identificó Salmonella sp en huevos procedentes de diferentes mercados de la ciudad de La Paz, mediante la técnica de Reacción en Cadena de la Polimerasa anidado o nested-PCR. De igual forma, se evaluó la sensibilidad y especificidad del método, comparada con la técnica convencional, para su posterior aplicación.
MATERIALES Y MÉTODOS
Control positivo
Se utilizó una cepa de Salmonella typhimurium ATCC 14028, facilitada por el Instituto de Investigaciones Fármaco Bioquímicas (IIFB) de la Facultad de Ciencias Farmacéuticas y Bioquímicas; y cepas de Salmonella sp aisladas de muestras clínicas del Instituto de Servicios de Laboratorio de Diagnóstico e Investigación en Salud (SELADIS). Se realizó la siembra de la cepas en medios de cultivo selectivos y diferenciales. Se aislaron colonias de Salmonella typhimurium y Salmonella sp en fase estacionaria en agar S-S (agar Salmonella-Shigella), se realizó con la extracción de ADN de Salmonella.
ExtracciónA los tubos Eppendorf se añadió PBS, se resuspendieron las colonias de Salmonella, se agitó, se centrifugó a 3000 rpm durante dos minutos, se retiró el sobrenadante y resuspendió con PBS: se centrifugó a 12000 r.pm. por dos minutos. Se desechó el sobrenadante y resuspendió con agua estéril, se incubó a 100° C (en aceite para lograr llegar a esa temperatura), se mezcló por pipeteo, se centrifugó a 12000 rpm durante dos minutos, se transfirió el sobrenadante a un tubo estéril y congeló a 4ºC. Al siguiente día, se descongelaron las muestras y se realizó la electroforesis.(25)
Optimización del Nested PCR
Posición y productos, producidos por los cebadores usados en la detección de Salmonella por PCR. (25)
Se preparó una primera mezcla de reacción utilizando los primers BR-SalA y BR-SalB, para identificar las secuencias SEQ No.:1 y 2; y una segunda mezcla utilizando los primers BR-Sal IA y BR-Sal IB, para identificar las secuencias SEQ No.:3 y 4 (tabla 3).
Los primers provienen del fragmento de 1.8 Kb HindIII de ADN cromosómico de Salmonella typhimurium (tabla 1).
Se utilizó el ADN extraído de la cepa control (Salmonella sp), se preparó el master mix o mezcla de reactivos PCR (tabla 2).
Se irradió con luz ultravioleta los materiales a utilizar; tips, gradillas, tubos, micropipetas, gorro, guantes y mangas. Se reconstituyó los primeros (tabla 3), para Salmonella sp, con agua estéril. Se diluyó los primers ya reconstituidos, también con agua estéril.
A la mezcla de reactivos PCR contenida en tubos PCR, se agregó las muestras y se amplificó en dos rounds en un termociclador (Termociclador PE 9600), previamente programado (tabla 4). Se reveló la electroforesis de ADN en transiluminador ultra violeta.
Optimización de extracción de ADN de Salmonella sp de muestras
Se marcaron los tubos, se añadió luego 700 ml de muestra y volumen igual de buffer de lisis (10 mM de tris, 10 mM de EDTA, 100 mM de NaCl y SDS 1%), 20 ml de proteinasa K, se mezcló, se incubó a 56°C durante cinco horas en Baño María con agitador, se centrifugó a 14000 rpm, se transfirió el sobrenadante a otro tubo, se añadió igual volumen de fenol cloroformo isoamilico, se centrifugó, se transfirió sobrenadante a otro tubo, se añadió igual volumen de solución SEVAG (cloroformo isoamilico 24:1), se mezcló, se centrifugó, se transfirió el sobrenadante a otro tubo, se añadió un décimo del volumen tomado de NaCl 3M, se agitó, y el doble de etanol absoluto frio, se dejó en reposo diez minutos, se centrifugó 15 minutos, se desechó el sobrenadante y secó a 60°C. Se realizó electroforesis. (25).
Aislamiento e identificación de Salmonella sp.
Se prepararon las muestras lavando los huevos con agua jabonosa, se enjuagó y sumergió en alcohol 70 % diez minutos, se extrajo el contenido a recipientes estériles,y se mezclo 25 ml de muestra en 225 ml de agua peptonada tamponada y se incubó a 35°C 24 horas. Se sembró en caldo tetrationato y caldo Rappaport-Vassiliadis, incubó a 42°C 24 horas. Se sembró en agar Salmonella-Shigella y agar xilosa-lisina-desoxicolato, incubando durante a 35°C 24 horas. Se sembrarón dos colonias en agar hierro triple azúcar, agar lisina hierro, SIM, caldo urea y agar citrato de Simmons.
Se utilizaron las mismas muestras y se extrajo ADN de la muestras, se amplificó las muestras de ADN extraído, se realizó la electroforesis y revelado.
RESULTADOS
Se logró extraer ADN de Salmonella typhimurium ATCC, 14028 y Salmonella sp, de las colonias aisladas en agar SS; ADN que fue utilizado como control positivo. Luego del revelado de la electroforesis del ADN extraído y amplificado, se obtuvieron bandas de ADN de 282 pares de bases (figuras 1, 2 y 3), que corresponden a Salmonella sp, producto de los primers BRSal1A y BRSal1B. Los primers BRSalA y BRSalB actuaron en el primer round.
Como se observa en la figura 1, se sembró en el pocillo 1 marcador de peso molecular, en el pocillo 2 agua del cuarto blanco (donde se realiza el master mix), en el pocillo 3 agua del cuarto donde se realizó la extracción de ADN, en el pocillo 4 control negativo (Shigella sp), en el pocillo 5 control positivo (Salmonella sp) y en los pocillos 6 y 7 muestras de ADN extraído. Se puede observar luego de electroforesis una banda de 282 pb (pares de bases) que corresponde al control positivo.
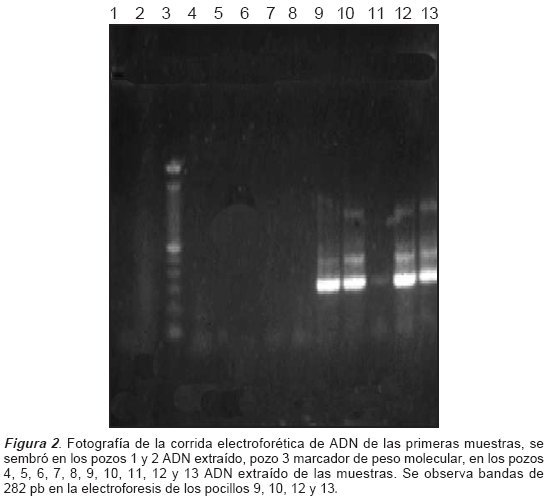
Como se observa en la Figura 2, se sembró en el pocillo 1 y 2 ADN extraído de las muestras en el pocillo 3 marcador de peso molecular, en los pocillos 4, 5, 6, 7, 8, 9, 10, 11, 12 y 13 ADN extraído de las muestras. Se observan en la electroforesis de los pocillos 9, 10 12 y 13, bandas de 282 pb.
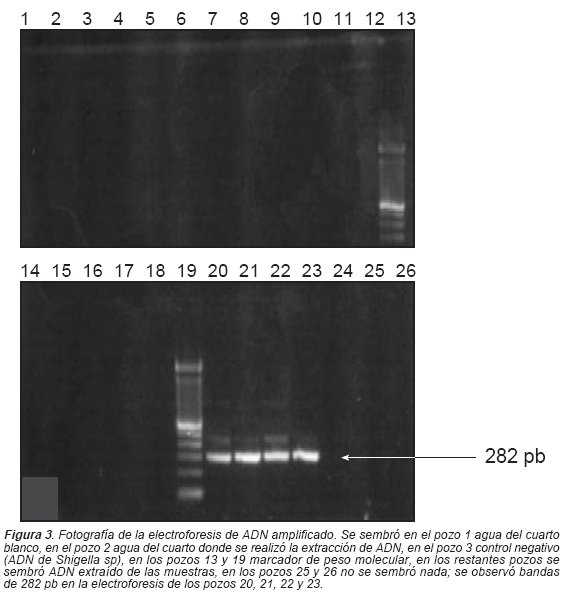
Como se observa en la Figura 3, se sembró en el pocillo 1 agua del cuarto blanco, en el pocillo 2 agua del cuarto donde se realizó la extracción de ADN , en el pocillo 3 control negativo (ADN de Shigella sp), en los pocillos 13 y 19 marcador de peso molecular, en los pocillos 4, 5, 6, 7, 8, 9, 10, 11, 12, 14, 15, 16, 20, 21, 22, 23 y 24 se sembró ADN extraído de las muestras, en los restantes pocillos no se sembró nada; también se observaron bandas de 282 pb en la electroforesis de los pocillos 20, 21, 22 y 23.
DISCUSIÓN
Los métodos utilizados para detectar estos patógenos fueron diseñados inicialmente para las muestras clínicas, algunos organismos en muestras alimentarias no tienen suficiente tiempo para producir colonias visibles en el medio, el método utilizado, Nested PCR, puede detectar 10 UFC (unidades formadoras de colonias). (25)
No se aisló Salmonella sp de cultivos, posiblemente por encontrarse en muy poca cantidad en los huevos para poder formar UFC.
Utilizando la técnica Nested PCR, se obtuvo los resultados, en el 17.5% de la muestras estudiadas, en la revelación de las corridas electroforéticas de las muestras de ADN extraídas. Se observa en las figuras 2 y 3; 8 muestras con bandas de 282 pb correspondientes a ADN de Salmonella sp.
La ventaja del uso de Nested PCR es la sensibilidad, ya que se utiliza una porción más pequeña de la muestra para obtener una amplificación específica y eficiente.(25)
Este estudio podría demostrar que el uso de las técnicas moleculares, tendría una ventaja sobre las técnicas convencionales si hablamos de tiempo, sensibilidad y especificidad. Pudiendo asi recomendar, esta técnica para la detección temprana de patógenos para el hombre. Si bien se requiere 105 bacterias de Salmonella para causar enfermedad, la sola presencia de Salmonella en los alimentos constituye un peligro para la salud.
AGRADECIMIENTO
Al Instituto de Servicios de Laboratorio de Diagnóstico e Investigación en Salud (SELADIS) por su apoyo a la investigación y en especial por el apoyo para este trabajo. Agradezco también al Dr. Giovanni García Rada y a los que participaron en la revisión y observaciones que fueron muy importantes para poder publicar este artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. Andreus, W., Manual of food quality control, 4 rev. Microbiological analysis, FAO, Washington, 1992, ISBN 92-5-10 3189-4. [ Links ]
2. Astiasaran, Yciar, Alimentos composición y propiedades, 2000. [ Links ]
3. Basualdo, Juan A., et al, Microbiología Biomédica, Ed Atlante srl, Buenos Aires, ISBN 950-9539-30-9.
4. Beller, Paloma, et al, Epidemiología de la salmonelosis no tifoidea en un hospital de Pontevedra, Enfermedades infecciosas y microbiología clínica, vol.18 No 5, mayo 2000.
5. Brock, Biología de los microorganismos, 8 edic., Ed. Pretince Hall, España, 1998. [ Links ]
6. Brooks, Geo F, et al, Microbiología Médica de Jawetz, 21 a edic., Ed. El Manual Moderno; México D.F.; 1999, ISBN 968-426-810-6. [ Links ]
7. CAE (1975) Capítulo XIV Huevos y derivados (3.14.00) Sección 1 huevos, Sección 2 derivados. [ Links ]
8. Calva, Edmundo, et al, Salmonella typhi y la fiebre tifoidea: de la biología molecular a la salud pública, Instituto de Biotecnología, UNAM. [ Links ]
9. Chen, Wilfred, Molecular Beacons: A Real-Time Polymerase Chain Reaction Assay for Detecting salmonella, Analytical Biochemistry, 2000. [ Links ]
10. Chin, James, et al, Publicación Científica y Técnica NO 581 El control de las enfermedades transmisibles, 17 edic., Informe oficial de la Asociación Estadounidense de Salud Pública, OPS. [ Links ]
11. Frazier, W. C., et al, Microbiología de los alimentos, 4 edic. Ed. Acribia S.A., Zaragoza - España, 1993. [ Links ]
12. Harris, Eva, et al, Alow-cost approach teo PCR: Appropriate transfer of biomolecular techniques, Ed. Oxford University Press Inc. Nueva York, 1998. [ Links ]
13. ICMSF: International Commission on Microbiological Specifications for Foods, Microorganismos de los alimentos 1: técnicas de análisis microbiológico, Ed. Acribia-Zaragoza, España. [ Links ]
14. Juklick D., Wolfgang, Microbiología de Zinsser, 20 edic.Ed. Panamericana, 1994. [ Links ]
15. Koneman, Elmer W., Diagnóstico Microbiológico, 5 edic., Ed. Panamericana, Buenos Aires, 1999, ISBN 950-06-1250-x. [ Links ]
16. Mac Faddin, Jean F., et al, Pruebas bioquímicas para la identificación de bacterias de importancia clínica, Ed. Médica Panamericana, México, 1993, ISBN 968- 7257-10-0. [ Links ]
17. Nesled PCR. [ Links ]
18. Pascual A., Ma Del Rosario, Microbiología alimentaria, Ed. Diaz de Santos, S.A., Madrid, España, 1992, ISBN 84-7978-030-4. [ Links ]
19. Rivas R., Virginia, Comparación de las técnicas de electroforesis en gel de poliacrilamida (PAGE), ELISA y Latex en la detección de rotavirus en heces fecales de niños menores de 5 años, Tesina para optar al título de licenciatura en bioquímica, La Paz, Bolivia, 2001. [ Links ]
20. Ruiz, Montserrat, et al, Infecciones extraintestinales producido por serotipos no tifoideos de Salmonella, Enfermedades infecciosas y microbiología clínica, vol 18 No. 5, Mayo 2000. [ Links ]
21. Sánchez M., Rolando S., Guía de trabajos prácticos de biología molecular, 1 edic., La Paz, Bolivia, 1998. [ Links ]
22. SEDES (Ministerio de Salud), Informe mensual de Laboratorio. Salmonelosis, ámbito urbano/rural, 2001. [ Links ]
23. Spiegel, Murray R., et al, Estadística: serie Schaum, Ed. Mc Graw-Hill. [ Links ]
24. US575332: Detection and identification of Salmonella and Shigella, artículo de Internet. [ Links ]
26. Valle R., Fernando M., Obtención e identificación de perfiles genéticos de Mycobacterium tuberculosis mediante DRE-PCR y su aplicación en la epidemiología molecular de la tuberculosis, Tesis de grado para la licenciatura en Bioquímica, La Paz, Bolivia, 1999. [ Links ]