Servicios Personalizados
Articulo
Indicadores
Links relacionados
 Citado por SciELO
Citado por SciELO
 Similares en SciELO
Similares en SciELO
Bookmark
BIOFARBO
versión impresa ISSN 1813-5363
BIOFARBO v.20 n.1 La Paz jun. 2012
ARTÍCULO ORIGINAL
Análisis de marcadores moleculares para la tipificación de Leishmania spp. circulantes en el departamento de La Paz, Bolivia
Analysis of molecular markers for typing of Leishmania spp. parasites circulating in La Paz department, Bolivia
Óscar V. Cárdenas Alegría1, Danitza Romero Calle1, Efraín Salamanca Capusiri1, José A. Santalla Vargas2, Patricia Oporto Portugal2, Daniela Arteaga Voigt3, María Teresa Alvarez Aliaga1, Enrique Terrazas Siles ()1, Ninoska Flores 1, Alberto Giménez Turba1
1 Instituto de Investigaciones Fármaco Bioquímicas – IIFB, Universidad Mayor de San Andrés. La Paz, Bolivia.
2Instituto Nacional de Laboratorios en Salud – INLASA, Ministerio de Salud y Deportes. La Paz, Bolivia.
3Instituto de Investigaciones Técnico Científicas de la Policía Boliviana. La Paz, Bolivia.
Dirección para correspondencia: Alberto Giménez Turba Ph.D. Av. Saavedra No 2224, Miraflores, 2ndo Piso.
Telf: 261-2431
E mail: agimenez@megalink.com
Recibido para publicación en: 26/04/12
Aceptado en: 27/06/12
RESUMEN
La identificación y diferenciación de especies de Leishmania es compleja por la similitud genética entre ellas, sin embargo el uso de técnicas moleculares ha facilitado este tipo de análisis. En el presente estudio se evalúo la capacidad de identificación y diferenciación de los complejos de Leishmania y Crithidia con distintos marcadores moleculares del Kinetoplasto: minicírculos (13A/13B), maxicírculos (Citocromo b) y la secuenciación del segmento cromosomal (7SL ARN). La amplificación del segmento del Citocromo b, permitió diferenciar entre los parásitos de los géneros Leishmania y Crithidia. La secuenciación del fragmento 7SL ARN permitió observar la similitud entres las secuencias de estos parásitos así como la construcción de árboles filogenéticos, que han permitido agrupar los parásitos del género Leishmania a nivel de subgénero y complejos, no obstante la diferenciación intra especie no fue posible. En base al análisis de secuenciación de 7SL ARN, de las nueve cepas de Leishmania aisladas a partir de pacientes procedentes de distintas regiones endémicas del departamento de La Paz, cinco cepas agruparon en el complejo Leishmania (V.) braziliensis y cuatro al complejo Leishmania (L.) mexicana. La utilización de este método molecular permite la diferenciación de las cepas circulantes de Leishmania, por lo que es una herramienta útil en la epidemiología de esta parasitosis.
Palabras Clave: Crithidia, Leishmania, tipificación, marcadores moleculares: 7SL ARN, Citocromo b, 13A/13B.
ABSTRACT
The identification and characterization of species of Leishmania parasites is complex, due to genetic similitude among them. However, molecular techniques have simplified this analysis. In this study, we evaluated the amplification of kinetoplast molecular markers amplified from minicircles (13A/13B), maxicircles (cytochrome b), and chromosomal sequencing (7SL RNA) using different strains of the Leishmania and Crithidia genera. The amplification of the cytochrome b segment differentiated the Leishmania and Crithidia genera. Genetic sequencing of the 7SL RNA fragment permitted observation of similarities between these parasites as well as phylogenetic tree construction, which made it possible to organize the Leishmania parasites at the subgenus and complex level, but no intra-species differentiation was found. The sequencing analysis of the nine strains of Leishmania, isolated from skin ulcers of patients from different endemic regions of the La Paz department in Bolivia, identified five strains among the Leishmania (V) braziliensis complex type and four strains within the Leishmania (L) mexicana complex type. The use of this molecular method allows the differentiation of the circulating strains of Leishmania parasites, and it is therefore a useful tool to update the epidemiology of this parasitosis
Key Words: Crithidia, Leishmania, typing, molecular markers; 13A/13B; 7SL RNA; Cytochrome b.
INTRODUCCIÓN
La leishmaniasis es un complejo de enfermedades infecciosas, de gran importancia epidemiológica y presenta múltiples manifestaciones clínicas (cutánea, muco-cutánea y visceral), causadas por protozoarios unicelulares del género Leishmania (amastigote y promastigote), transmitido al hombre mediante la picadura de dípteros de los géneros Phlebotomus y Lutzomyia. El género Leishmania está constituido por diversos complejos y especies las cuales pueden variar dependiendo de su ubicación geográfica. La manifestación clínica no sólo depende del genotipo del parásito, también del estado inmunológico del paciente y su genoma1,2,3. La identificación de las especies responsables de esta enfermedad permite determinar los brotes epidemiológicos, la distribución en regiones geográficas y predecir los futuros cambios en la transmisión de la enfermedad en un área determinada4.
La estructura genética poblacional de Leishmania tiene un análisis complejo, porque muchas especies presentan una limitada variabilidad genética en comparación con otras especies. La carga genética presente en los parásitos del orden Kinetoplastidae (cromosómico, kADN maxicírculos y minicírculos) es heterogénia, estas variaciones de carga genética se observa sobre todo entre especies de Leishmania pertenecientes al viejo y al nuevo mundo5,6,7.
Por otro lado, se ha reportado la presencia de diferentes parásitos con morfología heterogénea dentro de un insecto, en consecuencia dada la infección original del vector al huésped existiría la inoculación de distintos tipos de parásitos, en proporciones diferentes y con características propias de estos8. Por tal razón, el aislamiento de los parásitos patógenos, a partir de los vectores, se ve influenciada por cultivos mixtos9, la toma de muestra a partir pacientes puede también corresponder a una mezcla de diferentes parásitos y de genotipos10,11.
Crithidia sp. es un parásito no patógeno para el humano, es utilizado para el análisis de especificidad y sensibilidad de distintos marcadores moleculares, por la estrecha relación genética que tiene con Leishmania12,13. Marcadores moleculares como el miniexon, ADN ribosomal son capaces de diferenciar Leishmania de otros flagelados no patógenos, géneros como Endotrypanum y Crithidia14 y son considerados como "grupo externo" en el análisis filogenético, asimismo Crithidia sp. es un modelo para estudios genéticos comparativos evolutivos con Leishmania9.
Los parásitos de estos géneros presentan diferencias morfológicas que no son fáciles de diferenciar al microscopio óptico, como los choanomastigotes son similar a los promastigotes de Leishmania15; estos parásitos no patógenos pueden interferir en los cultivos de Leishmania y Trypanosoma, Crithidia sp. tiene la capacidad de sobrevivir en agua entre 30 a 48 horas por lo que tienen mayor éxito de ser aislados en los cultivos in vitro16.
La tipificación de Leishmania es generalmente realizada por isoenzimas siendo considerada como una prueba estándar para la caracterización de estas cepas17,18. En los últimos tiempos el avance de la tecnología ha permitido mejorar la identificación y caracterización de este parásito, especialmente en los métodos moleculares, así como la amplificación mediante la reacción en cadena de la polimerasa (PCR) de segmentos conservados de los minicírculos (kADN), maxicírculos y otros19,20,21. La utilización de la secuenciación de estos segmentos amplificados permite mejorar la tipificación, asimismo se observa el polimorfismo de simples nucleótidos (SNPs) de las distintas secuencias genéticas utilizadas, que constituyen una información útil en la tipificación de especies y análisis filogenético de Leishmania sp22. En el presente estudio se realizó la amplificación por PCR para el análisis de los marcadores moleculares de minicírculos (13A/13B), maxicírculo (citocromo b) y la secuenciación de un segmento cromosómico 7SL ARN para la tipificación de Leishmania23.
MATERIAL Y MÉTODOS
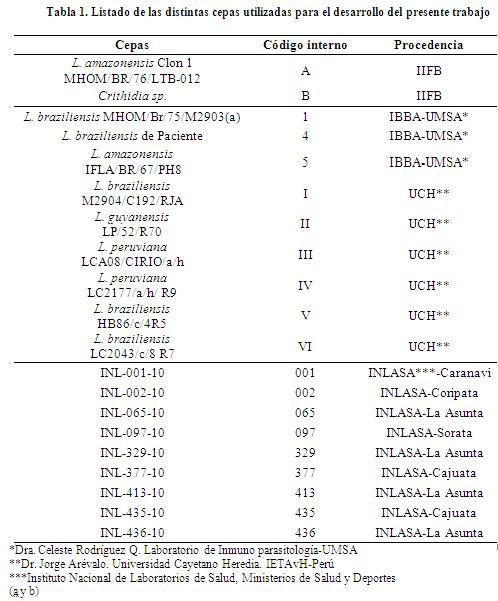
Cepas. El presente trabajo tiene un diseño metodológico detallado en el Esquema 1 y se utilizaron 21 cepas (1 cepa de Crithidia y 20 cepas de Leishmania). De ellas, un total de 17 cepas fueron adaptadas a cultivo in vitro en el IIFB. Entre las cultivadas, 6 cepas de Leishmania fueron donadas por el Dr. Jorge Arévalo, de la Universidad Cayetano Heredia, a través del Instituto de Enfermedades Tropicales Alexander von Humboldt, Lima, Perú. Un total de 8 cepas de Leishmania, congeladas desde el año 1994, fueron donadas por la Dra. Celeste Rodríguez del Laboratorio de Inmuno-parasitología del IBBA, Facultad de Medicina de la UMSA de las cuales solamente 4 cepas fueron utilizadas en este trabajo y además 9 cepas de Leishmania fueron aisladas a partir de úlceras cutáneas de pacientes provenientes de distintas regiones endémicas del departamento de La Paz, en el Laboratorio de Parasitología del INLASA (Tabla 1).
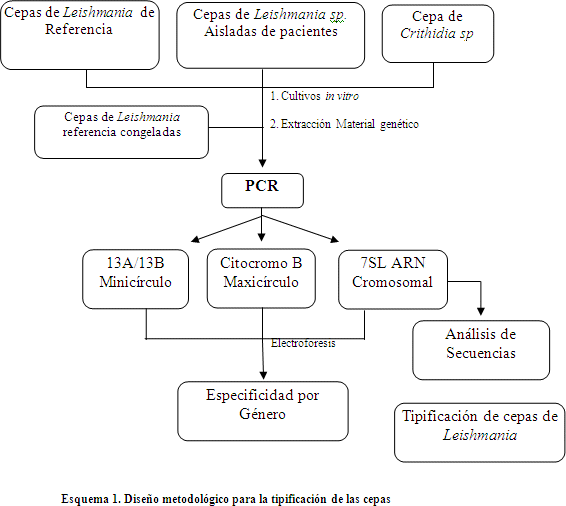
Cultivo de los parásitos. Las cepas I al VI, a y b fueron adaptadas a cultivo in vitro en medio Schneider (pH 6,8) suplementado con Glutamina, CaCl2 6% (p/v), Bicarbonato de Sodio 40% (p/v), Gentamicina 200 mg/L y 5% de suero albumino bovino (los reactivos utilizados fueron adquiridos de SIGMA-Aldrich). Los cultivos fueron conservados en una estufa a 27º C24. El mantenimiento de los cultivos fue vigilado, observando el movimiento y saturación de los parásitos a diario, a través del empleo de un microscopio óptico invertido (Zeiss, Axiovert 25). Para la observación de las características morfológicas se prepararon frotis y tinciones de Giemsa, para posteriormente ser observados al microscopio.
Extracción del material genético. Para la extracción del material genético se aplico el método de precipitado por salado modificado23, los cultivos de parásitos (~2mL) fueron centrifugados a 7.000 rpm por 10 minutos, se adicionó al precipitado 300µL de una solución de lisis (Guanidina 5M, EDTA 20mM, Tris-HCl pH 7,0, 40mM y Tween 20, 1%), se mezcló con ayuda de un vortex (Thermolyne). Esta suspensión fue incubada a 65º C por 15 minutos y posteriormente se agregó 200µL de una solución fría de acetato de sodio (3M, pH 7,5), a continuación se mezcló por inmersión. Las suspensiones fueron centrifugadas a 13.000 rpm por 15 minutos, el sobrenadante se trasvasó a un nuevo tubo, se adicionó etanol absoluto frío, los tubos con el sobrenadante fueron incubadas en hielo por 30 minutos y posteriormente se centrifugaron a 13.000 rpm por 10 minutos. El precipitado fue lavado con etanol al 70% y la suspensión fue nuevamente centrifugada a 13.000 rpm por 1 minuto, repitiendo este último procedimiento tres veces. El precipitado fue secado a 55º C por 15 minutos, se resuspendió con 100µL del tampón TE (pH 8, EDTA 1mM, Tris-HCl 10 mM pH 7) y finalmente fue conservado a 4 ºC hasta su utilización. La calidad del material genético obtenido fue evaluada a través de la observación por la electroforesis horizontal, se utilizó geles de agarosa (1%) teñidos con Bromuro de Etidio (0,5 µg/mL; PROMEGA) y se visualizó con ayuda de un transiluminador de luz ultravioleta (Spectroline). La cantidad de ADN fue determinada con las lecturas de absorbancia (260, 280 y 320 nm) con un espectrofotómetro Cintra 5 (GBC Scientific Equipment).
Reacción en cadena de la polimerasa (PCR). Se utilizó el material genético en aquellas muestras que presentaban valores del índice de pureza dentro lo esperado (1,65 a 1,85)23 y se realizó la optimización para los diferentes marcadores, así como el kinetoplasto: de minicírculos, de maxicírculos y ADN cromosómico del parásito. Los cebadores utilizados (IDT, USA) y las condiciones optimizadas en un termociclador Eppendorf de las distintas PCRs se detallan en la Tabla 2; los productos obtenidos de las diferentes cepas evaluadas en el presente trabajo fueron observados mediante electroforesis horizontal en geles de agarosa (2%), teñidos con Bromuro de Etidio (0,5 μg/mL; PROMEGA) y visualizados con un transiluminador a luz ultravioleta (Ultraline). Se utilizó un marcador de peso molecular de 0,1 a 1,5 kpb (100bp DNA ladder, PROMEGA).
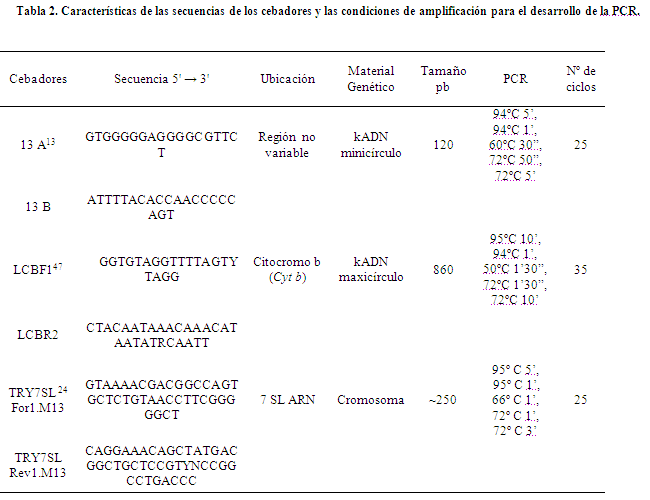
Secuenciación de los productos de PCR fragmento 7SL ARN. Los productos obtenidos de la amplificación del segmento 7SL ARN, fueron sometidos a la purificación y amplificación, se utilizaron los protocolos de Applied Biosystems para la secuenciación. Los cebadores utilizados para la reacción de secuenciación del fragmento 7SL ARN fueron: M13 Forward, 5-GTAAAACGACGGCCAG-3 y M13 Reverse, 5-CAGGAAACAGCTATGAC-324 y la secuenciación fue realizada en un analizador genético ABI 3130 Genetic Analyzer (Applied Biosystems).
Análisis de las secuencias. Los productos obtenidos de las secuencias forward y reverse de cada muestra fueron alineadas con la secuencia de referencia de L. tarantolae (Nº de Acceso GenBank AY781796.1) a través del programa BioEdit Sequence Aligment Editor25. Se verificaron los electroferogramas de los sitios variables en las secuencias analizadas y se editaron las secuencias para obtener secuencias consenso. Se identificó el porcentaje de similitud con las secuencias depositadas en el banco de genes, para esto se utilizó el nucleotide BLAST (Basic Local Alignment Search Tool), del National Center for Biotechnology Information (NCBI) en la página web www.ncbi.nlm.nih.gov.
Se elaboró un fichero de texto con las secuencias obtenidas del fragmento del gen 7SL ARN y las depositadas en el GenBank de las diferentes especies de Leishmania, Crithidia y de T. brucei, se utilizó el método CLUSTAL W para el múltiple alineamiento con el programa BioEdit. Para la construcción del árbol filogenético se utilizó el programa MEGA 426.
RESULTADOS
Se realizó el cultivo axénico de los diferentes parásitos de Leishmania sp. y Crithidia sp. en laboratorios del IIFB, ambos géneros de parásitos fueron cultivados en similares condiciones de crecimiento, obteniéndose a las 72 horas abundante cantidad de promastigotes y choanomastigotes de Leishmania sp. y Crithidia sp., respectivamente. En el caso del cultivo de choanomastigotes de Crithidia sp. se observó una mejor adaptación al medio de cultivo. Al mismo tiempo se observó en los cultivos de Crithidia la formación de una fina película (biofilm), que no se presenta en los cultivos de Leishmania.
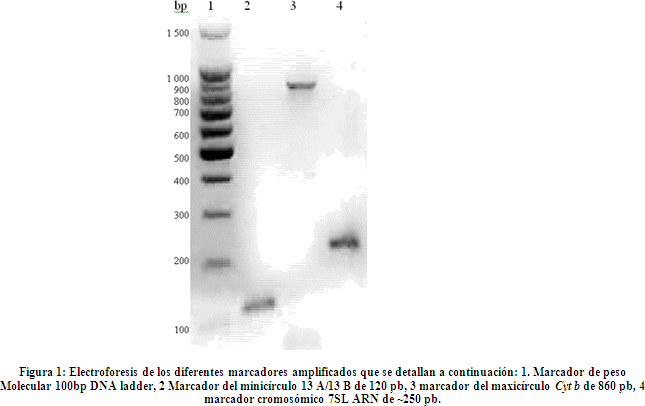
A los diferentes cultivos se aplicó el método de extracción de precipitado por salado y se obtuvo aproximadamente 300 µg/ml del material genético a partir de ~106 parásitos por mililitro de cultivo. Todas las cepas presentaron valores promedio de 1,7 del índice de pureza, sin embargo se excluyeron cuatro cepas del estudio provenientes del IBBA, debido a que los valores del índice de pureza de estas cepas no estaban dentro lo esperado, considerándose como material genético de pobre calidad. Se optimizaron los protocolos de la PCR para los diferentes marcadores 13A / 13B, Cyt b y 7SL RNA, obteniéndose los productos en los tamaños esperados de cada marcador (Figura 1), Posteriormente se evaluaron las distintas cepas de Leishmania sp, y de Crithidia sp. con cada uno de los diferentes marcadores, encontrándose que solamente el marcador del maxicírculo Cyt b, logró diferenciar al género Leishmania sp. de Crithidia sp. (Tabla 3).

Los productos secuenciados del fragmento 7SL ARN de las distintas cepas de Leishmania y de Crithidia fueron analizados con los programas descritos anteriormente, se encontró la diferenciación de los géneros, subgénero y se logró agrupar en los distintos complejos a los parásitos estudiados. Es así que las secuencias de las cepas I al VI, 1 y 4 presentaron una alta similitud (99%) con las especies del complejo L. (V.) braziliensis (L. braziliensis, L. panamensis) y la cepa II fue identificada como L. guyanensis (Figura 2).

Las secuencias del segmento 7SL ARN pertenecientes a las cepas a y 5 presentaron semejanza con L. amazonenesis y por lo tanto estas secuencias agruparon con las diferentes especies del sub-género Leishmania pertenecientes al complejo de L. (L.) mexicana (Figura 2). Con respecto a la cepa b la secuencia presentó un 99% de similitud con Crithidia sp. La presencia de dos especies C. fasciculata y C. luciliae en este género se diferencian entre estas por la variación nucleotídica en la posición 127; para C. fasciculata la presencia de una guanina (G), y una adenina (A) para C. luciliae en esa posición (Figura 2), la cepa b una semejanza con la especie de C. fasciculata. El polimorfismo de los nucleótidos del fragmento 7SL ARN está presente en distintas posiciones, estas variaciones nucleotídicas son particulares para cada especie parasitaria; asimismo, en las posiciones 100 hasta la 150 existe mayor variación, aparentemente este sitio pertenece a un dominio de la molécula 7SL ARN27, estas variaciones permitió la construcción del árbol filogenético (Figura 3), en donde, se observa la agruparon de los distintos complejos de Leishmania, género Crithidia y la diferenciación de algunas especies de cada complejo. La aplicación del método de secuenciación del segmento 7SL ARN, realizando la tipificación de 9 cepas aisladas a partir de úlceras cutáneas se observó que 5 de las 9 cepas agruparon en el complejo de L. (V.) braziliensis (muco-cutánea) y el restante de las cepas (4 de 9) al complejo de L. (L.) mexicana (cutánea), ver Figura 3, encontrándose valores de similitud de 98 a 100%.

DISCUSIÓN
En el desarrollo del trabajo se realizó el cultivo de parásitos de choanomastigotes de Crithidia y promastigotes de Leishmania en el medio de Shneider presentan similares morfologías, sin embargo se observaron diferencias entre estos parásitos, es así, Crithidia sp tiene la capacidad de realizar la formación de biofilm que se adhiere a la superficie de las cajas de cultivo27, principalmente en los cultivos mantenidos por tiempos prolongados (más de 72 horas). Los parásitos del género Crithidia sp. son fáciles de cultivar, tienen un crecimiento acelerado, no es indispensable la utilización de suero en el medio de cultivo y no se requiere precauciones especiales de bioseguridad en el manipuleo en contraste a Leishmnia sp 9,28.
Con respecto a la obtención del material genético la cantidad y calidad dependió de la conservación de las cepas, el factor tiempo y la forma de conservación de los microorganismos podrían influenciar en la obtención del material genético extraído, por otro lado es necesario considerar para una mejor identificación y/o caracterización de microorganismos patógenos la cantidad, calidad y tipo del material genético a analizar, genético del núcleo (cromosomal), del kinetoplasto (maxicírculo, minicírculos).
El análisis mediante la PCR de los distintos marcadores 13A/13B, Cytb y 7SL RNA, se encontró que solamente el marcador del maxicírculo Cytb logró diferenciar a nivel de género; aunque los cebadores 13A/13B son ampliamente utilizados para la identificación de Leishmania en muestras de pacientes, hospederos intermediarios y vectores en diferentes estudios13,29,30 que amplifican segmentos conservados de los minicírculos, se ha reportado la existencia de reacción cruzada con la región intergénica del cromosoma 8 de humanos (Homo sapiens NCBI–Nº Accession: AC026765)31. Por otro lado, el mecanismo de replicación y segregación a las células hijas de los minicírculos en el kinetoplasto es aleatorio, por lo tanto la nueva descendencia podría s er diferente una de otra32; además no todos los segmentos de los minicírculos son útiles para la determinación por PCR debido al bajo número de copias de los mismos, lo que influiría en la identificación y caracterización de los parásitos aislados33, el mantenimiento de cultivos a largo plazo constituiría uno de los principales factores que propician este tipo de variaciones y afecta también a la virulencia de Leishmania34. Por otra parte es necesario indicar la similitud genética y fenotípica que presenta Crithidia con Leishmania35,36,37,38, sobre todo en las secuencias de los minicírculos y podrían haber influenciado en el resultado obtenido por la PCR. Con respecto al análisis del marcador de maxicírculo Cytb de Leishmania ha permitido constituir un potencial marcador para la identificación de este parásito y de otros no patógenos al humano, permite también la diferenciación intraespecie en el caso que se realice la secuenciación de este marcador39,40,41,42, estableciéndose como un marcador útil para la identificación, diferenciación y análisis filogenético de Leishmania sp, no se encontró reacción cruzada con el material genético humano43, no obstante, es importante considerar la heteroplasmía presente en el kinetoplasto del parásito 44,45,46.
Los productos amplificados del marcador cromosómico 7SL ARN en las cepas de Leishmania sp. y Crithidia sp. fueron sometidos a la secuenciación, la utilización de este marcador fue considerada para la identificación por secuenciación de Leishmania sp. por presentar abundante copias de este gen en el genoma del parásito, existen dominios conservados del 7SL RNA entre los componentes de los Trypanosomatidae, la similitud intra-especie de las secuencias (81 a 100%) permite identificar al parásito a nivel de género, distinguir los complejos y especies de Leishmania24, la evaluación de las distintas cepas utilizadas en este estudio mediante la secuenciación permitió la identificación y la diferenciación de estas. Con relación a la identificación de las seis cepas (I al VI) concedidas por el Instituto de Enfermedades Tropicales Alexander von Humbolt de la Universidad Cayetano Heredia, Lima Perú, ellas fueron identificadas con los marcadores isomerasa manosa fosfato (MPI), cistein proteinasa B (CPB) y la proteína de choque térmico 70 (HSP70), se determinó que las cepas I, V y VI son L. brazileinsis, las cepas III y IV son L. peruviana y la cepa II se trata de la especie L. guyanensis, de acuerdo las indicaciones realizadas por el Dr. Jorge Arevalo. Además se logró la caracterización de cepas a nivel de especie, sin embargo en las cepas III y IV identificadas como L. peruviana no fueron identificadas a este nivel, ya que en la base de datos del 7SL ARN no existe la secuencia particular de esta especie; en el segmento analizado de las cepas presentan similares secuencias entre las especies de L. braziliensis y L. panamensis, en la secuencia de L. guyanenesis presenta una variación en la posición 160 de timina (T) por citosina (C) que es particular de esta especie.
La variación genética encontrada entre los complejos de Leishmania dificulta la identificación y diferenciación de las distintas especies patógenas de Leishmania. Estudios comparativos entre los genomas de Leishmania identificaron pocos genes específicos propios de algunas especies (L. major, L. infantum, L. brazilensis, aunque estas especies se han separando hace 15 a 50 millones de años). Por otro lado es necesario considerar la búsqueda de un marcador molecular permita analizar a los clones de las poblaciones en una misma especie11,47. Las poblaciones clonales del parásito esta relacionada con el tipo de reproducción de Leishmania48, por otra parte, es importante el intercambio genético que sufren estos parásitos49,50, el intercambio genético es crucial para la sobrevivencia del parásito. Los híbridos pueden permitir la adaptación a nuevos nichos ecológicos, vectores y hospederos incluyendo humanos y animales domésticos49,50. La diversidad de las cepas en las distintas áreas geográficas refleja la plasticidad de estos parásitos al adaptarse a diferentes especies de vectores (o poblaciones de estos); la incorporación de marcadores moleculares robustos podría permitir mejorar la determinación de la diversidad genética de varias especies de Leishmania que circulan en la naturaleza51,52.
La utilidad de métodos moleculares tiene mucha importancia en los estudios de epidemiología de esta enfermedad en Bolivia, por lo tanto es importante considerar la utilización de distintos métodos moleculares, además del uso de cepas no patógenas similares genéticamente a Leishmania para la evaluación de marcadores moleculares utilizados en la identificación y diferenciación de este parásito.
AGRADECIMIENTOS
A los proyectos: UMSA-ASDI-SAREC: Enfermedades Infecciosas, UMSA/IDH-2009. Desparasitación de Niños en Escuelas Rurales, UMSA-ASDI 2009 Valoración Química y Biológica de Extractos Acuosos de la especie Galipea longiflora y el proyecto AECI A/024457/09 de Francisco Bolás Fernández, (UCM) y Ma. Auxiliadora Dea Control de la Leishmaniosis en Bolivia: Evaluación in vitro de la sensibilidad/resistencia de cepas autóctonas de Leishmania spp. a los tratamientos convencionales, por parte del financiamiento. A las instituciones: IRD por ofrecer la beca para la Maestría en Ciencias Biológicas y Biomédicas de la UMSA. Instituto Nacional de Laboratorios de Salud (INLASA), la Facultad de Ciencias Farmacéuticas y Bioquímicas (FCFB) de la UMSA y el Instituto de Investigaciones Técnico Científicas de la Policía Boliviana por el apoyo en el uso del secuenciador de la FCFB.
REFERENCIAS
1. Dedet J P, Pratlong Fr, Lanotte G, Ravel C. The Parasite. Clin in Dermatology 1999;17:261–268. [ Links ]
2. Hide M B, Kamhawi S, Bras-Gon R, Sundar S and Bañuls A L. Understanding Human Leishmaniasis: The Need for an Integrated Approach. Ency Infec Diseases 2007;87-123.
3. Lipoldová M, Demant P. Genetic susceptibility to infectious disease: lessons from mouse models of leishmaniasis. Nature 2006;7: 294-305. [ Links ]
4. Volpini C, Marques M J, Lopes S S, Machado C G, Mayrink W and Romanha A J. Leishmania identification by PCR of Giemsa-stained lesion imprint slides stored for up to 36 years. European Soc Clin Microbiol Infec Diseases 2006;12(8):815–818.
5. Souza W de, Attias M, Rodrigues J C F. Review: Particularities of mitochondrial structure in parasitic protists (Apicomplexa and Kinetoplastida). The Int J Biochem & Cel Biol. 2009; 41: 2069–2080.
6. Peacock C S, Seeger K, Harris D, Murphy, Ruiz Jeronimo C, Quail M A., Peters N, Adlem E, Tivey A, Aslett M, Kerhornou A, Ivens A, Fraser A, Rajandream M A, Carver T, Norbertczak H, Chillingworth T, Hance Z, Jagels K, Moule S, Ormond D, Rutter S, Squares R, Whitehead S, Rabbinowitsch E, Arrowsmith C, White B, Thurston S, Bringaud F, Baldauf S L., Faulconbridge A, Jeffares D, Depledge D P., Oyola S O., Hilley J D, Brito L O, Tosi L R O, Barrell B, Cruz A K, Mottram J C, Smith D F & Berriman M. Comparative genomic analysis of three Leishmania species that cause diverse human disease. Nat Genetics 2007;39(7):839-847. [ Links ]
7. Vallejo G. Estudio comparative entre las secuencias de kDNA de Trypanosoma cruzi y Trypanosoma rangeli y su aplicación en el diagnostico Molecular de la tripanosomiasis Americana. Actual. Biol1998;20(68):43-56. [ Links ]
8. Yurchenko V Y, Lukes J, Jirku M, Maslov. Selective recovery of the cultivation-prone components from mixed trypanosomatid infections: a case of several novel species isolated from Neotropical Heteroptera. Int J Syst and Evol Microbiol 2009;59:893-909.
9. Podlipaev Sergei A. Insect Trypanosomatids: the Need to Know More. Mem Inst Oswaldo Cruz 2000; 95(4): 517-522. [ Links ]
10. Bañuls A.L., Hide M., Tibayrenc M. Molecular epidemiology and evolutionary genetics of Leishmania parasites. Int J Parásitol 1999; 29:1137-1147. [ Links ]
11. Criscione C, Poulin R and Blouin M. Molecular ecology of parasites: elucidating ecological and microevolutionary processes. Mol Ecol 2005;14:2247-2257.
12. Morales M A., Cruz I, Rubio J M, Chicharro C, Cañavate C, Laguna F and Alvar J. Relapses versus Reinfections in Patients Coinfected with Leishmania infantum and Human Immunodeficiency Virus Type 1. J Infec Diseases 2002; 185: 1533–7.
13. Deborggraeve S, Laurent T, Espinosa D, Van der Auwera G, Mbuchi M, Wasunna M, El-Safi S, Almustafa Al-Basheer A, Arévalo J, Miranda-Verástegui C, Leclipteux T, Mertens P, Dujardin J C, Herdewijn P and Büscher P. A Simplified and Standardized Polymerase Chain Reaction Format for the Diagnosis of Leishmaniasis. J Infec Diseases 2008;198:1565–72.
14. Paiva B R, Secundino N F C, Nascimento J C, Pimenta P.F.P., Galati E.A.B., Andrade J H.F., Malafronte R.S. Detection and identification of Leishmania species in field-captured phlebotomine sandflies based on mini-exon gene PCR. Acta Tropic 2006;99:252–259. [ Links ]
15. Harris E, Kropp G, Belli A, Rodriguez B and Agabian N. Single-Step Multiplex PCR Assay for Characterization of New World Leishmania Complexes. J of Clin Microbiol1998;36(7):1989–1995.
16. Buck G, Berriman M, Donelson J, El-Sayed N, Kissinger J, Simpson L, Tait A, Teixeira M and Beverley S. Pathogenomics of Trypanosomatid parasites. White Paper 2007;1-33.
17. Spanakos G, Piperaki E T, Menounos P G., Tegos N Flemetakis A, Vakalis N C. Detection and species identification of Old World Leishmania in clinical samples using a PCR-based method. Trop Med Hyg 2008; 102:46-53. [ Links ]
18. Asgari Q, Hossein MM, Mehrabani D, Oryan A, Reza H G, Mohammad O S and Paykari H. Zoonotic cutaneous leishmaniasis in Shiraz, Southern Iran: A molecular, isoenzyme and morphologic approach. J Res Med Sc 2007; 12(1)7-15.
19. Castilho TM., Shaw J,Floeter-Winter LM. New PCR Assay Using Glucose-6-Phosphate Dehydrogenase for Identification of Leishmania Species. J Clin Microbiol 2003;41(2):540–546. [ Links ]
20. Chocholová E, Jirků M, Luke J. A diagnostic assay based on variable Intergenic Region distinguishes between Leishmania donovani and Leishmania infantum. Folia Parasitol 2008;55:75–78. [ Links ]
21. Cárdenas OA. Análisis de los marcadores genéticos de minicírculos (13 A/13 B), maxicírculos (Cyt B) y cromosómico (7 SL ARN) para la identificación y diferenciación molecular de especies de Leishmania spp. a partir de cultivos in vitro . UMSA; 2011. [ Links ]
22. Sereno D, Semestre J. Use of an enzymatic micromethod to quantify amastigote stage of Leishmania amazonensis in vitro. Parasitol.Res.1997;83:401-403. [ Links ]
23. Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids. Sym Ser 1999; 41:95-98 [ Links ]
24. Tamura K, Dubley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol 2009;24:1596-1599. [ Links ]
25. Zelazny AM, Fedorko DP, Neva LFA., et al. Evaluation of 7SL RNA gene sequences for the identification of Leishmania spp. Am. J. Trop. Med. Hyg 2005;72(4): 415–420 [ Links ]
26. Oliveira LMF, Soares MJ, Souza W. Ultrastructure of the Adhesion of Crithidia guilhermei (Kinetoplastida: Trypanosomatidae) to Plastic Coverslips: Formation of Fibrous Matrix Around the Cells. Mem Inst Oswaldo Cruz 1997;92(4):549-552. [ Links ]
27. Calderón AO, Chinchilla M, Avendaño A, et al.Cinética multiplicativa de clones de Crithidia fasciculata (Kinetoplastida: Trypanosomatidae) en un sistema de cultivo in Vitro. Parásitol. Latinoam 2006;61:32–36. [ Links ]
28. Bensoussan E, Nasereddin A, Jonas F, et al.Comparison of PCR Assays for Diagnosis of Cutaneous Leishmaniasis. J Clin Microbiol 2006;44(4):1435–1439.
29. Lachaud L, Marchergui HS, Chabbert E, et al. Comparison of Six PCR Methods Using Peripheral Blood for Detection of Canine Visceral Leishmaniasis. J Clin Microbiol 2002 ; 40(1): 210–215 [ Links ]
30. Vergel C, Walker J, Saravia NG. Amplifiction of human DNA by primers targeted to Leishmania Kinetoplast DNA and post-genome consideration in the detection of parasites by a polymerase chain reaction. Am. J. Trop. Med. Hyg 2005;72(4):423–429. [ Links ]
31. Simpson L, Thiemann OH., Savilli N, et al. Evolution of RNA editing in trypanosome mitochondria. Proc. Natl. Acad. Sci 2000;97(13):6986-6993. [ Links ]
32. Lambson B, Barker DC. Basic molecular tools: Homologous minicircles in Leishmania donovani. Trop Med Hyg 2002;96:75-180. [ Links ]
33. Pandey K, Yanagi T, Dev PB, et al. Characterization of Leishmania isolates from Nepalese patients with visceral leishmaniasis. Parasitol Res. 2007;100:1361–1369. [ Links ]
34. Fernandes AP, Nelson K, Beverley SM. Evolution of nuclear ribosomal RNAs in kinetoplastid protozoa: Perspectives on the age and origins of parasitism (RNA editing/mitochondrial rRNA/Bodo caudaus/Trypanosomatidae). Proc. Natl. Acad. Sci 1993;90:11608-11612. [ Links ]
35. Moreira D, López G, Vickerman K. An update view of kinetoplastid phylogeny using enviromental sequences and a closer outgroup: proposal for a new classification of the class Kinetoplastea. Int J Sys Evol Microbiol 2004;54:1861-1875. [ Links ]
36. Maslov DA, Podlipaev SA, Luke J. Phylogeny of the Kinetoplastida: Taxonomic Problems and Insights into the Evolution of Parasitism. Mem Inst Oswaldo Cruz 2001;96(3):397-402. [ Links ]
37. Stothard JR. Future Trypanosomatid Phylogenies: Refined Homologies, Supertrees and Networks. Mem Inst Oswaldo Cruz 2000;95(4):523-526. [ Links ]
38. Marco JD, Uezato H, Mimori T, et al. Are cytochrome b gene sequencing and polymorphism-specific polymerase chain reaction as reliable as multilocus Enzyme Electrophoresis for identifyin Leismania spp. From Argentina?. Am J Trop Med Hyg. 2006;75(2):256–260. [ Links ]
39. Luyo AG, Uezato H, Oshiro M, et al. Sequence variation of the Cytochrome b gene of various human infecting members of the genus Leishmania and their phylogeny. Parasitol 2004; 128: 483–491. [ Links ]
40. Foulet F, Botterel F, Buffet P, et al. Detection and Identification of Leishmania Species from Clinical Specimens by Using a Real-Time PCR Assay and Sequencing of the Cytochrome b Gene. J Clin Microbiol 2007;45(7):2110–2115. [ Links ]
41. Asato Y, Oshiro M, Kaung MC, et al. Phylogenic analysis of the genus Leishmania by cytochrome b gene sequencing. Exp Parasitol 2009;121:352–361. [ Links ]
42. Myint CK, Asato Y, Yamamoto Y, et al. Polymorphisms of cytochrome b gene in Leishmania parasites an d their relation to types of cutaneous leishmaniasis in Pakistan. Jap Dermatol Ass 2008;35:76-85. [ Links ]
43. Flegontov PN, Strelkova MV, Kolesnikov AA. The Leishmania major maxicircle divergent region is variable in different isolates and cell types. Mole & Bioch Parásitol 2006;146:173–179. [ Links ]
44. Flegontov PN, Zhirenkina EN, Gerasimova ES, et al. Selective amplification of maxicircle classes during the life cycle of Leishmania major. Mol & Bioch Parásitol 2009;165:142–152. [ Links ]
45. Lynn MA, McMaster WR. Leishmania: conserved evolution – diverse diseases. Trends Parásitol 2008;24:103-105. [ Links ]
46. Rougeron V, DeMeeus T, Hide M, Etienne W, et al. Extreme inbreeding in Leishmania brazilensis. PNAS 2009;106(25):1024-1029. [ Links ]
47. Akopyants NS, Kimblin N, Secundino N, Patrick R, Peters N, et al. Demonstration of Genetic Exchange During Cyclical Development of Leishmania in the Sand Fly Vector. Science 2009;324:265-268. [ Links ]
48. Hide M, Bañuls AL.Species-specific PCR assay for L. infantum/L. donovani discrimination. Act Tropica 2006;100:241–245. [ Links ]
49. Miles MA, Yeo M, Mauricio IL. Leishmania Exploit Sex. Science 2009;324:187-188. [ Links ]
50. Calvopina M, Armijos RX, Marco JD, Uezato H, Kato H, et al. Leishmania isoenzyme polymorphisms in Ecuador: Relationships with geographic distribution and clinical presentation. BMC Infec Diseases 2006;6:1-9. [ Links ]
51. Rodríguez GI, Marín C, Vargas F, Córdova O, et al. Identication and biochemical characterization of Leishmania strains isolated in Peru, Mexico, and Spain. Exp Parásitol 2006;112:44–51. [ Links ]
52. Cupolillo E, Regina BL, Toaldo CB, et al. Genetic Polymorphism and Molecular Epidemiology of Leishmania (Viannia) braziliensis from Different Hosts and Geographic Areas in Brazil. J Clin Microbiol 2003;41(7):3126–3132.